Актуальні теми
#
Bonk Eco continues to show strength amid $USELESS rally
#
Pump.fun to raise $1B token sale, traders speculating on airdrop
#
Boop.Fun leading the way with a new launchpad on Solana.
Що потрібно комп'ютеру, щоб вивчити правила сполучення основ РНК?
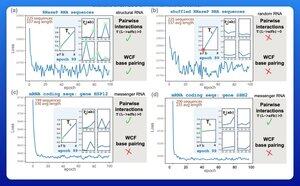
Люди тренують великі мовні моделі для прогнозування структури РНК. Деякі з цих моделей мають сотні мільйонів параметрів.
Захоплюючим раннім результатом стало те, що ці моделі вивчають правила сполучення баз Watson-Crick-Franklin безпосередньо на основі даних.
Дослідницька група з Гарварду вирішила з'ясувати, яка найменша можлива модель може досягти такого результату.
Вони тренували крихітну імовірнісну модель лише з 21 параметром за допомогою градієнтного спуску.
При наявності всього 50 послідовностей РНК - без відповідних структур - правила сполучення основ вискочать вже через кілька тренувальних епох.
Отже, відповідь на їхнє початкове запитання полягала в тому, що для вивчення цього типу моделі потрібно «набагато менше, ніж ви думаєте».
Я не думаю, що це означає, що масштабні тренувальні зусилля обов'язково дурні або помилкові. Але цей результат свідчить про те, що існує багато ефективності та продуктивності, які все ще можна отримати від інновацій в архітектурі.
У мові біології є багато глибинних структур.
3,6K
Найкращі
Рейтинг
Вибране













